python 读取DICOM头文件的实例
用dicompyler软件打开dicom图像,头文件如图所示:
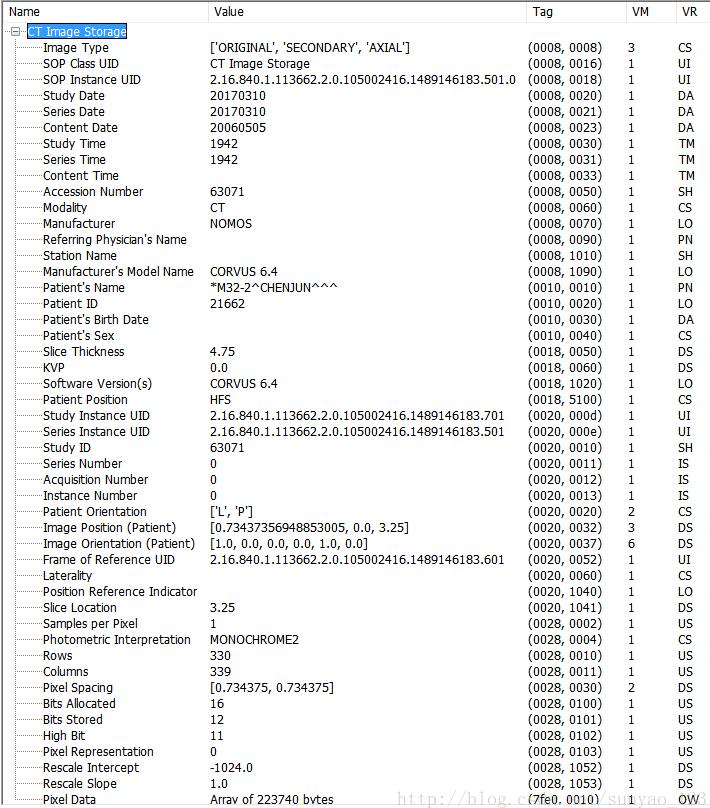
当然也可以直接读取:
ds = dicom.read_file('H:\Data\data\\21662\\2.16.840.1.113662.2.0.105002416.1489146183.701\CT\\CT#0#21662#E7AB693D.dcm')
print ds
>>
(0008, 0008) Image Type CS: ['ORIGINAL', 'SECONDARY', 'AXIAL']
(0008, 0016) SOP Class UID UI: CT Image Storage
(0008, 0018) SOP Instance UID UI: 2.16.840.1.113662.2.0.105002416.1489146183.501.0
(0008, 0020) Study Date DA: '20170310'
(0008, 0021) Series Date DA: '20170310'
(0008, 0023) Content Date DA: '20060505'
(0008, 0030) Study Time TM: '1942'
(0008, 0031) Series Time TM: '1942'
(0008, 0033) Content Time TM: ''
(0008, 0050) Accession Number SH: '63071'
(0008, 0060) Modality CS: 'CT'
(0008, 0070) Manufacturer LO: 'NOMOS'
(0008, 0090) Referring Physician's Name PN: ''
(0008, 1010) Station Name SH: ''
(0008, 1090) Manufacturer's Model Name LO: 'CORVUS 6.4'
(0010, 0010) Patient's Name PN: '*M32-2^CHENJUN^^^'
(0010, 0020) Patient ID LO: '21662'
(0010, 0030) Patient's Birth Date DA: ''
(0010, 0040) Patient's Sex CS: ''
(0018, 0050) Slice Thickness DS: '4.75'
(0018, 0060) KVP DS: '0'
(0018, 1020) Software Version(s) LO: 'CORVUS 6.4'
(0018, 5100) Patient Position CS: 'HFS'
(0020, 000d) Study Instance UID UI: 2.16.840.1.113662.2.0.105002416.1489146183.701
(0020, 000e) Series Instance UID UI: 2.16.840.1.113662.2.0.105002416.1489146183.501
(0020, 0010) Study ID SH: '63071'
(0020, 0011) Series Number IS: '0'
(0020, 0012) Acquisition Number IS: '0'
(0020, 0013) Instance Number IS: '0'
(0020, 0020) Patient Orientation CS: ['L', 'P']
(0020, 0032) Image Position (Patient) DS: ['0.73437356948853', '0', '3.25']
(0020, 0037) Image Orientation (Patient) DS: ['1', '0', '0', '0', '1', '0']
(0020, 0052) Frame of Reference UID UI: 2.16.840.1.113662.2.0.105002416.1489146183.601
(0020, 0060) Laterality CS: ''
(0020, 1040) Position Reference Indicator LO: ''
(0020, 1041) Slice Location DS: '3.25'
(0028, 0002) Samples per Pixel US: 1
(0028, 0004) Photometric Interpretation CS: 'MONOCHROME2'
(0028, 0010) Rows US: 330
(0028, 0011) Columns US: 339
(0028, 0030) Pixel Spacing DS: ['0.734375', '0.734375']
(0028, 0100) Bits Allocated US: 16
(0028, 0101) Bits Stored US: 12
(0028, 0102) High Bit US: 11
(0028, 0103) Pixel Representation US: 0
(0028, 1052) Rescale Intercept DS: '-1024'
(0028, 1053) Rescale Slope DS: '1'
(7fe0, 0010) Pixel Data
发现一套图的第一张和最后一张的Slice Thickness往往和中间层的值是不同的。
Path = 'H:\Data\data\\21662\\2.16.840.1.113662.2.0.105002416.1489146183.701\CT\\a.dcm' slices = dicom.read_file(path) spacing = slices.PixelSpacing
读取头文件信息方法:
Slices.上图头文件中的name列。
这时要注意的是,name大小写不变,去掉空格,去掉符号,比如括号。
举例:
origin = slices.SoftwareVersions print origin >>CORVUS 6.4
spacing = slices[1].PixelSpacing print spacing >>['0.734375', '0.734375']
以上这篇python 读取DICOM头文件的实例就是小编分享给大家的全部内容了,希望能给大家一个参考,也希望大家多多支持【听图阁-专注于Python设计】。